SIMINDの使い方3 ~PyTomographyによる再構成 OSEM & OSMAPOSL & BSREM~

※SIMINDの使い方を説明している動画(YouTube)には、以下のようなものがあります。ぜひ、合わせて勉強してみてください。
1. Online Conference SIMINDの基本的な使用方法
2. Online Conference CT画像からデジタルファントムを作成する。
3. SIMIND Monte Carlo SPECT Simulator: Part 1
4. SIMIND Monte Carlo SPECT Simulator: Part 2
5. SIMIND Monte Carlo SPECT Simulator: Part 3
6. SIMIND Monte Carlo – Part 1
7. SIMIND Monte Carlo Part 2
8. SIMIND Monte Carlo – Part 3
9. SIMIND Monte Carlo – Part 4
SIMIND入力ファイル(tools4radtech.smc)
SIMINDのシミュレーション条件を記載したsmcファイルは以下のものになります。セキュリティの問題から拡張子を.smc.txtにしていますので、使用する際には.txtは削除してください。
加えて、使用したボクセルファントム(mumap.dmi)とボクセルソース(source.smi)は以下のものです。2つのファイルは128x128x128のマトリクスで16-bit unsigned、little endianで保存します。
mumap.dmiは画素値が1000(CT画像の画素値0に+1000したもの)なので、水を意味します。
source.smiは大きな球の画素値が50で、内部の小さな球の画素値を200としています。


tools4radtech.smc.txtをtools4radtech.smcとしたあと、こちらのページで開いて(main-5)、内容を確認しましょう。
以下のように設定しています。
Flag-5:TrueーSPECT撮像をする
Flag-14:Trueー投影データのファイルにInterfile ver.3.3形式のヘッダー情報を含める。
Flag-15:TrueーSPECT画像のpixelサイズおよびスライス厚に再サンプリングされたファントムを保存する。
Main-11:mumapーmumap.dmiを使う
Main-12:sourceーsource.smiを使う
Index-14:-1ーボクセルファントムを使う
Index-15:-1ーボクセルソースを使う
Index-29:120ーSPECTのprojection数
Index-34:128ーdensity mapのスライス枚数
Index-76:256ーprojection imageの列数
Index-77:256ーprojection imageの行数
Index-78:128ーdensity mapのI方向(列)のマトリクスサイズ
Index-81:128ーdensity mapのJ方向(行)のマトリクスサイズ
Index-79:128ーsource mapのI方向(列)のマトリクスサイズ
Index-82:128ーsource mapのJ方向(行)のマトリクスサイズ
Index-84:0ーダミールーチン
※Index-28は0.12くらいにした方が良かったかも
Index-28=プロジェクション画像の画素サイズ [cm/pixel]
また、実行する際には以下のようにコマンドを打ちました。
cd C:\simind #SIMINDのフォルダに移動
simind tools4radtech /CA:1 /IN:x22,3x /NN:3 /PX:0.234 /TH:0.234このコマンドはtools4radtech.smcファイルを入力として実行し、
/CA:1ー保存する情報(air, tot, scaの3種類)を指定
/IN:x22,3xーsimind.iniの22番目の項目を3に変更(Flag-15で保存される画像の画素値をmuにする)
/NN:3ー試行回数を3倍にする。試行回数=ボクセルソースの画素値の合計×投影方向
/PX:0.234ーボクセルソースの画素サイズを0.234cmx0.234cmにする
/TH:0.234ーボクセルソースのスライス厚を0.234cmにする。
実行中の画面は以下の通りです。
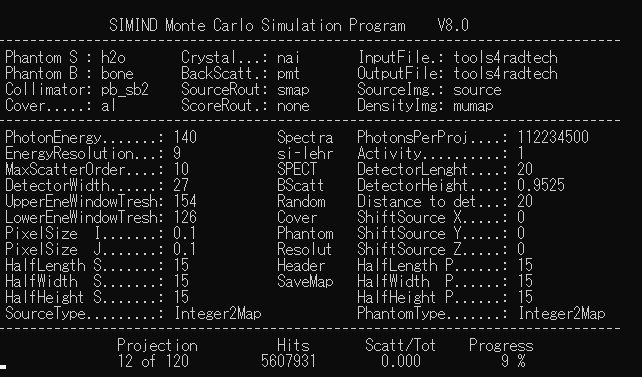
計算が終わると以下のようなファイルが出力されているのが分かります。
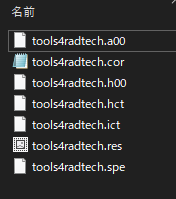
それぞれのファイルは以下の通り。
.a00ープロジェクションデータ
.h00ープロジェクションデータ(ヘッダーファイル)
.corー投影角度の情報
.ictーFlag-15によって出力された画像(画素値がmuで、吸収補正に使う)
.hctーFlag-15によって出力された画像(ヘッダーファイル)(画素値がmuで、吸収補正に使う)
.resー結果のログ
.speースペクトルデータ
このデータを使って、今度は再構成をします。
Pytomographyによる再構成
最初にも言った通り、このサイトのままやっていきます。
実行する環境はGoogle colabなどがいいでしょう。準備等はググってください。
また、pytomographyのインストールも必要なのでpipなどで入れておいてください。インストールガイドも参照してください。
作業フォルダをsimindフォルダにするか、先ほどの出力ファイルをどこかに移して、そこを作業フォルダにするかは任意です。
from __future__ import annotations
import os
from pytomography.io.SPECT import simind
from pytomography.projectors.SPECT import SPECTSystemMatrix
from pytomography.transforms.SPECT import SPECTAttenuationTransform, SPECTPSFTransform
from pytomography.algorithms import OSEM, OSMAPOSL, BSREM, KEM
import matplotlib.pyplot as plt
import torch
from pytomography.priors import RelativeDifferencePrior
from pytomography.priors import TopNAnatomyNeighbourWeight
from pytomography.likelihoods import PoissonLogLikelihood
from pytomography.transforms.shared import KEMTransform
from pytomography.projectors.shared import KEMSystemMatrix
import os
もしこの段階でエラーなどが出ていれば、必要なライブラリ・モジュールをインストールしてください。
print(os.getcwd())このコマンドで、現在の作業フォルダを確認することができます。
photopeak_path = 'c:\\simind\\tools4radtech.h00'
attenuation_path = 'c:\\simind\\tools4radtech.hct'photopeak_pathという変数の中にプロジェクションデータのヘッダーファイル(*.h00)、attenuation_pathという変数の中にμマップのヘッダーファイル(.hct)のパス(文字列)を入れます。windowsだと\\でエスケープしたりと、少し書き方が難しいですね。
photopeak = simind.get_projections(photopeak_path)
print(photopeak.shape)photopeakという変数の中にprojectionデータを格納します。
printするとtorch.Size([120, 256, 256])と表示されました。120方向のデータで、それぞれ256×256のマトリクスです。torchという形式で読み込まれているようですね。
plt.figure(figsize=(5,4))
plt.pcolormesh(photopeak[0].cpu().T, cmap='magma')
plt.axis('off')
plt.colorbar(label = 'Counts per second per MBq')
plt.show()いったん、photopeakの中を見てみましょう。
最初の投影[0]を表示します。
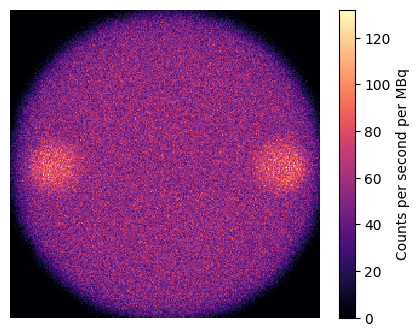
こんな感じで表示されました。少し横長になっているのはfigsize=(5,4)を適当に指定しているからです。
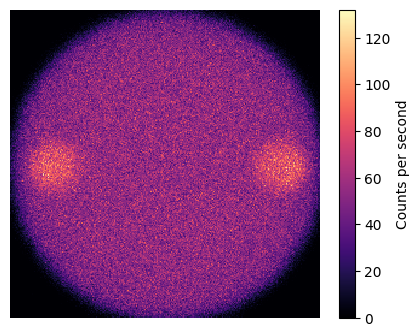
dT = 99999 # seconds per projection
photopeak_realization = torch.poisson(photopeak * dT)
photopeak = photopeak_realization次に投影データに収集時間とポアソンノイズを追加します。
simindの計算結果はcounts/MBq/secに規格化されています。
plt.figure(figsize=(5,4))
plt.pcolormesh(photopeak[0].cpu().T, cmap='magma')
plt.axis('off')
plt.colorbar(label = 'Counts per second')
plt.show()描画してみます。収集時間(dT = 99999)を長くしているのでノイズの加算はほとんどないです。
object_meta, proj_meta = simind.get_metadata(photopeak_path)projectionデータのメタ情報を取得します。それぞれobject_metaとproj_meta変数で格納します。
attenuation_map = simind.get_attenuation_map(attenuation_path)
att_transform = SPECTAttenuationTransform(attenuation_map)Flag-15で作成したμマップを読み込みます。attenuation_pathは最初のコードですでにパスを取得しています。
psf_meta = simind.get_psfmeta_from_header(photopeak_path)
psf_transform = SPECTPSFTransform(psf_meta)PSF情報を情報を取得します。
system_matrix = SPECTSystemMatrix(
obj2obj_transforms = [att_transform,psf_transform],
proj2proj_transforms = [], # PETで使用
object_meta = object_meta,
proj_meta = proj_meta
)システムマトリクスを作成します。検出行列はシステムマトリクスの一部に含まれ、その他減弱やPSFなどが組み込まれます。放射線源からの信号が検出器でどのように観測されるかをモデル化し、最終的にその観測データから元の画像を再構成するのに必要です。
likelihood = PoissonLogLikelihood(system_matrix, photopeak)ポアソン分布を過程した尤度関数を求めます。この関数のソースコードはこちらです。
OSEM
recon_algorithm = OSEM(likelihood)
recon_OSEM = recon_algorithm(n_iters = 4, n_subsets = 8)尤度関数の勾配(傾き)を利用して、画像の各ボクセルの値を少しずつ更新していき、尤度を最大化するoptimizerを設定します。まずはoptimizerとしてOSEMを使います。先ほどの尤度関数を渡します。
計算を実行します。今回はCPUを使っているので結構時間がかかりました(数時間)。
結果を表示します。
plt.subplots(1,2,figsize=(16,6))
plt.subplot(121)
plt.pcolormesh(recon_OSEM.cpu()[:,:,120].T, cmap='magma')
plt.colorbar()
plt.axis('off')
plt.title('Coronal Slice')
plt.subplot(122)
plt.pcolormesh(recon_OSEM.cpu()[70].T, cmap='magma')
plt.colorbar()
plt.axis('off')
plt.title('Sagittal Slice')
plt.show()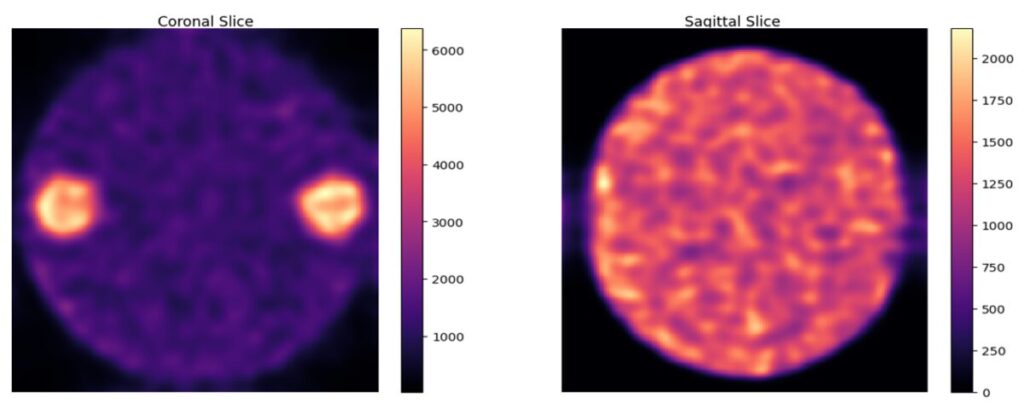
右側の図はホット球が映っていないsagittalの画像でバックグラウンド領域が明るくなっています。
想定した通りの画像ですね。
保存しておこうと思います。
import torch
import numpy as np
import tifffile
from torchvision import transforms
# tensor を NumPy 配列に変換
numpy_array = recon_OSEM.numpy()
numpy_array = np.transpose(numpy_array, (1, 2, 0))
# TIFF 形式で保存
tifffile.imwrite('tools4radtech_osem_ite4_sub8_120.tiff', numpy_array)OSMAPOSL
次はoptimizerにOSMAPOSLを使ってみます。
OSMAPOSLとはOS(EM)+MAP+OSLです。MAPなので事前情報を組み込むことになります。OSLはone step laterで、計算が効率化されるとかなんとか・・・(よく分かりません)
weight_top8anatomy = TopNAnatomyNeighbourWeight(attenuation_map, N_neighbours=8)
prior_rdpap = RelativeDifferencePrior(beta=0.3, gamma=2, weight=weight_top8anatomy)MAPの事前情報として、解剖学的な情報を組み込みます。解剖学的な情報としてmumapを使います。8近傍(近い8個のボクセル)の重みを求めます。重みは距離や構造の類似度から求めるようで、似たボクセルは近い値になるように調整されます。また、罰則関数にはRelativeDifferencePriorを使っています。βは正則化の強さを調整し、大きくするほど画像が滑らかになります。γは近接ボクセル間の差分のスケーリングファクターで、どの程度エッジを強調するかを決めます。
recon_algorithm_osmaposl = OSMAPOSL(
likelihood = likelihood,
prior = prior_rdpap)
recon_osmaposl = recon_algorithm_osmaposl(n_iters = 40, n_subsets = 8)それでは、尤度関数と罰則関数を渡します。
plt.subplots(1,2,figsize=(16,6))
plt.subplot(121)
plt.pcolormesh(recon_osmaposl.cpu()[:,:,120].T, cmap='magma')
plt.colorbar()
plt.axis('off')
plt.title('Coronal Slice')
plt.subplot(122)
plt.pcolormesh(recon_osmaposl.cpu()[70].T, cmap='magma')
plt.colorbar()
plt.axis('off')
plt.title('Sagittal Slice')
plt.show()
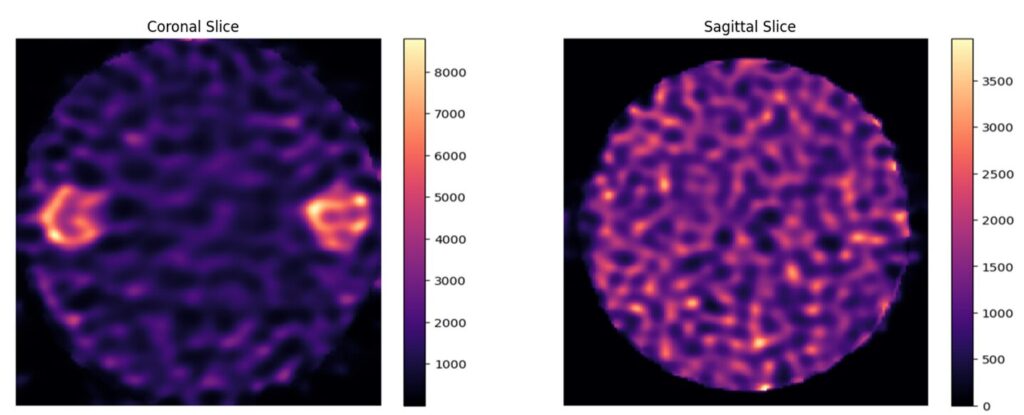
あれ?あんまり画質良くないですね
BSREM
GEが採用している方法で、Q.clearの中心的な役割を担っています。
先ほどのweight_top8anatomyとprior_rdpapを使います。
recon_algorithm_bsrem = BSREM(
likelihood = likelihood,
prior = prior_rdpap,
relaxation_sequence = lambda n: 1/(n/50+1)
)
recon_bsrem = recon_algorithm_bsrem(40,8)さっきとは違い、RAMLAの発展形であるBSREMには緩和パラメータもあります。
更新回数nが増加するほど、更新量は小さくなります。
計算に10時間かかりました・・・
plt.subplots(1,2,figsize=(16,6))
plt.subplot(121)
plt.pcolormesh(recon_bsrem.cpu()[:,:,120].T, cmap='magma')
plt.colorbar()
plt.axis('off')
plt.title('Coronal Slice')
plt.subplot(122)
plt.pcolormesh(recon_bsrem.cpu()[70].T, cmap='magma')
plt.colorbar()
plt.axis('off')
plt.title('Sagittal Slice')
plt.show()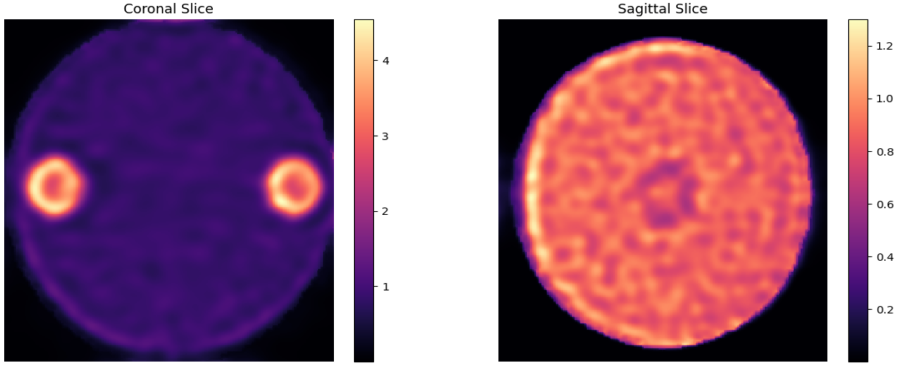
きれいな画像です。
今回は以上になります










有益な情報ありがとうございます。SIMIND初心者ですので大変勉強になります。
そこでSIMINDについて質問ですが、こちらのサイトのようにCT画像からImageJ でデジタルファントムを作成しましたが、SIMINDで以下のようなエラーが出てしまいます。
‘The source maps seem to contain no counts. Check the validity of the source map and then try again!’
確実にvalueは入っていますし、SIMINDでのパラメータも間違いないと思っていますが、このエラーの原因について、ご助言をお願いしたいです。何卒よろしくお願いいたします。
色々と試してみないとわかりませんが、リトル/ビッグエンディアン、16bit unsigned、コマンドライン引数の/PXと/THを正しく指定しているかなどはどうでしょうか。記事が書き間違えている可能性もあります。
お返事ありがとうございます。
ご指摘いただいた箇所を確認し、再度シミュレーションしたらうまくいきました!
ありがとうございます。